Twinning Projekt
- Ansprechperson:
- Projektbeteiligte:
Institut für biologische Grenzflächen 1 (IBG-1), KIT, Eggenstein-Leopoldshafen, Deutschland
- Starttermin:
11/2017
- Endtermin:
11/2020
Das Ziel des Projektes ist die Entwicklung eines neuartigen, computergestützten, kontinuierlichen Bio-Mikro-Reaktors mit Bestandteilen aus Nano- und Mikrometer Dimensionen. Eine enge Kooperation des Instituts für Mikroverfahrenstechnik (IMVT) mit dem Institut für Biologische Grenzflächen 1 (IBG-1) stellt in diesem Projekt den Grundsatz für die Verbindung von Modellierung bzw. Simulation (IMVT) und Experimenten (IBG-1).
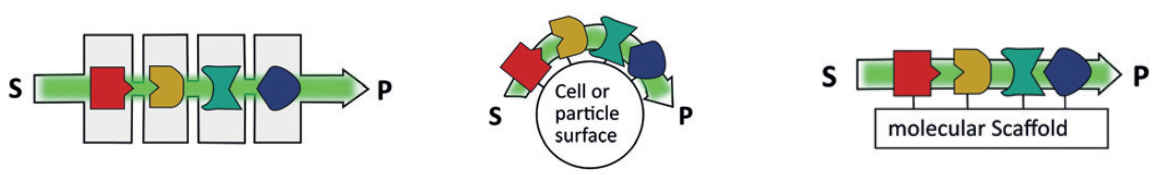
Die Zelle ist ein komplexer, natürlich vorkommender Bioreaktor, in dem viele, teilweise sich beeinflussende Reaktionsnetzwerke vorzufinden sind. Um solche Systeme besser zu verstehen, stellte sich die Separation und Unterteilung spezifischer Reaktionspfade und Reaktionen als eine vielversprechende Methode heraus. Realisiert wird dies durch eine räumliche Trennung der aktiven Enzyme und anschließender Kombination zu einer Multienzym-Kaskade. Eine solche Kaskade kann auf unterschiedlichste Weisen umgesetzt werden. Der Austrag von katalytischen Enzymen ist hierbei nicht erwünscht und wird durch eine Immobilisierung auf Hilfsoberflächen verhindert. Man unterscheidet zwischen separaten Reaktoren (a), multienzym-Kaskade auf Partikeln (b) oder DNA-Origami-Strukturen auf Partikeln (c) (vgl. Abbildung 1).
Abbildung 1: Reaktordesigns einer Multienzym-Kaskade. (a) sequentielle Enzym-Reaktoren; (b) immobilisiert auf einem Partikel; (c) immobilisiert auf einer DNA-Origami-Struktur. [1]
In dem Projekt soll mittels auf Partikeln immobilisierten Ketoreduktasen und einem Cofaktor Regenerationssystem ein Diketon stereoselektiv reduziert werden. In Bezug auf entsprechende Designs sollen Reaktormodelle erstellt und mit Experimenten validiert werden.
Mittels synthetischen, mit Enzymen modifizierten DNA-Origami-Strukturen können unterschiedliche Anordnungen von Enzymen experimentell untersucht werden. Molekulardynamische Simulationen sollen im Vorhinein potentielle, effizienzsteigernde Konfigurationen ermitteln und ermöglichen somit eine effiziente, ökonomische, experimentelle Versuchsplanung.
Der angestrebte Bioprozess soll mittels geeigneter Reaktor- und Kinetik-Modellen eine Vorhersage und Optimierung vom Reaktorverhalten ermöglichen. Dabei gilt es, neben makroskopischen Reaktor- und Partikelmaße, Fluideigenschaften und Reaktionsbedingungen auch den Einfluss der Enzymanordnungen auf DNA-Strukturen zu untersuchen.
[1] Rabe, K. S., Müller, J., Skoupi, M., & Niemeyer, C. M. (2017). Cascades in Compartments: En route to Machine‐Assisted Biotechnology. Angewandte Chemie International Edition.